
Magdalena Masłoń - Biologia RNA
Tematyka badawcza: Prawidłowe funkcjonowanie komórek wymaga precyzyjnej koordynacji ekspresji genów, a zaburzenia w jej regulacji leżą u podstaw wielu chorób rozwojowych. Nasza grupa bada mechanizmy ekspresji genów odpowiedzialne za rozwój embrionalny ssaków.
Etos: rzetelność naukowa, atmosfera szacunku i wsparcia
Narzędzia: mysie i ludzkie komórki macierzyste, modele mysie, sekwencjonowanie, edycja genomu za pomocą CRISPR-Cas9, screeny siRNA, bioinformatyka.
Podsumowanie naukowe:
Ekspresja informacji genetycznej podlega regulacji w odpowiedzi na różne czynniki środowiska. Na przykład, podczas rozwoju embrionalnego, prawidłowa ekspresja genów umożliwia powstanie tysięcy typów komórek z niewielkiej populacji komórek pluripotencjalnych.
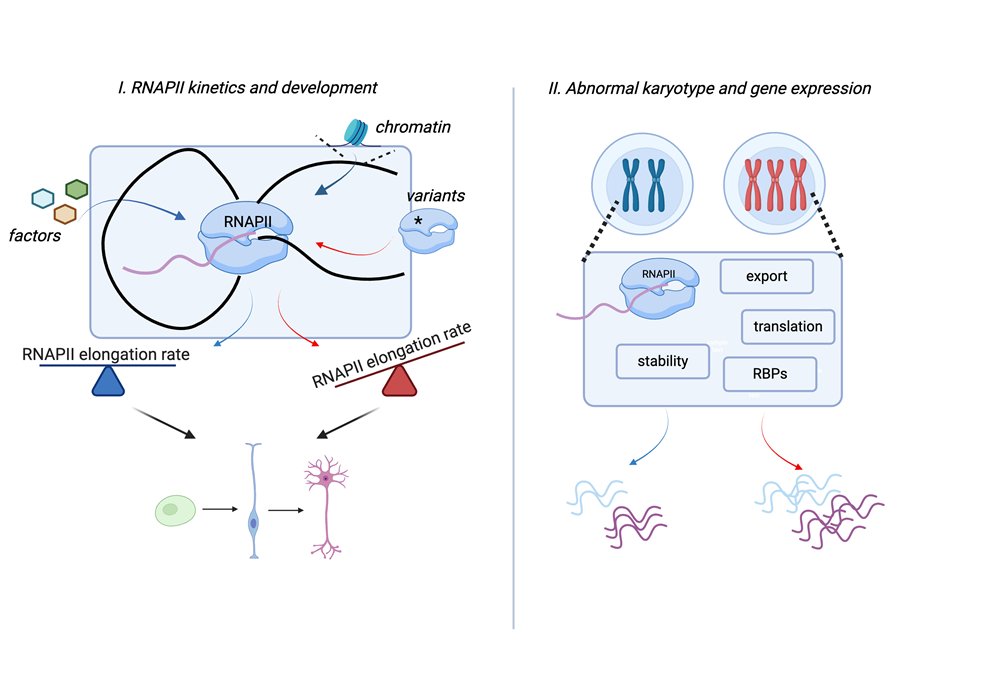
Polimeraza RNA II (RNAPII) jest enzymem odpowiedzialnym za transkrypcję genów, czyli proces w którym powstają wszystkie kodujące mRNA i niektóre niekodujące RNA. Prędkość transkypcji jest zmienna dla różnych genów i dla różnych części tego samego genu. Zmienna prędkość RNAPII wpływa na ilość wyprodukowanmego mRNA a także na proces dojrzewania pre-mRNA, a zatem ma fundamentalne znaczenie w regulacji ilości oraz rodzaju wytwarzanych białek.
W moich ostatnich badaniach stworzyłam model mysi RNAPII z mutacją punktową (Polr2aR749H), która powoduje spowolnienie prędkości RNAPII (tzw. "wolna" RNAPII). Wykazałam, że "wolna" RNAPII jest zmianą letalną na etapie rozwoju zarodkowego, a komórki embrionalne z “wolną” RNAPII nie różnicują się prawidłowo w komórki układu nerwowego. Na poziomie molekularnym, “wolna” polimeraza nie jest skuteczna w transkrypcji długich genów, które kodują białka niezbędne do prawidłowej funkcji neuronów. Te odkrycia sugerują, że kinetyka transkrypcji pełni istotną funkcję w rozwoju embrionalnym,. Co ciekawe mechanizmy odpowiedzialne za regulację transkrypcji podczas rozwoju oraz konsekwencje zaburzeń w jej regulacji, nie są w pełni poznane.
1 dzielone współautorstwo
Haward F1, Maslon MM1, Yeyati PL1, Bellora N, Hansen JN, Aitken S, Lawson J, von Kriegsheim A, Wachten D, Mill P, Adams IR, Caceres JF. Nucleo-cytoplasmic shuttling of splicing factor SRSF1 is required for development and cilia function. Elife. 2021 Aug 2;10:e65104. doi: 10.7554/eLife.65104. PMID: 34338635
Maslon MM, Braunschweig U, Aitken S, Mann AR, Kilanowski F, Hunter CJ, Blencowe BJ, Kornblihtt AR, Adams IR, Cáceres JF. A slow transcription rate causes embryonic lethality and perturbs kinetic coupling of neuronal genes. EMBO J. 2019 May 2;38(9):e101244. doi: 10.15252/embj.2018101244.
Maslon MM1, Heras SR1, Bellora N, Eyras E, Cáceres JF. The translational landscape of the splicing factor SRSF1 and its role in mitosis. Elife. 2014 May 6;3:e02028. doi: 10.7554/eLife.02028.

dr Magdalena Masłoń


